Cientistas identificam descoberta-chave no controle do crescimento vegetal
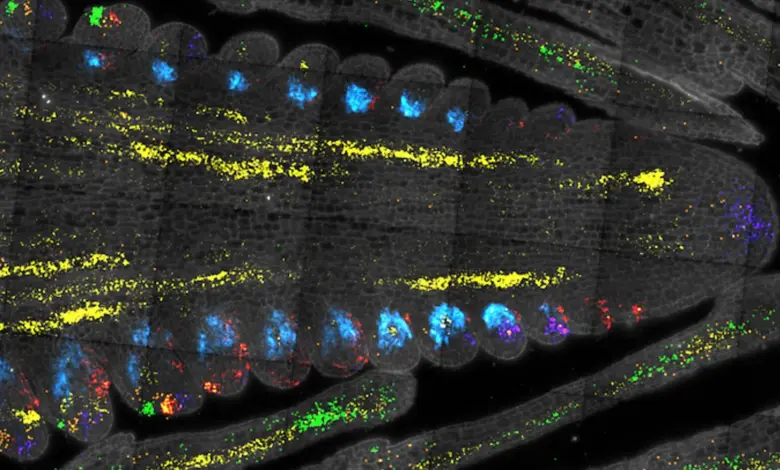
Cold Spring Harbor, Nova York — InkDesign News — Pesquisadores do Cold Spring Harbor Laboratory (CSHL), nos Estados Unidos, mapearam de forma inédita reguladores de células-tronco vegetais em milho e Arabidopsis, utilizando uma técnica avançada de sequenciamento de RNA em célula única. O estudo, publicado recentemente, pode orientar o desenvolvimento de culturas agrícolas mais produtivas e resilientes.
O Contexto da Pesquisa
Células-tronco vegetais sustentam a produção mundial de alimentos, rações e biocombustíveis, ao constituírem o núcleo central do crescimento das plantas. Apesar de sua importância, ainda existem lacunas consideráveis no entendimento sobre os mecanismos genéticos que controlam essas células essenciais. Estudos anteriores não conseguiram localizar diversos genes influentes na regulação dessas funções. A equipe do CSHL procurou superar esse desafio empregando tecnologias de ponta e associando diferentes espécies vegetais.
Resultados e Metodologia
O grupo concentrou-se inicialmente em dois conhecidos reguladores de células-tronco, denominados CLAVATA3 e WUSCHEL. O trabalho começou com a dissecação minuciosa de pequenos brotos de milho e Arabidopsis, etapa conduzida pela pesquisadora Xiaosa Xu, ex-pós-doutoranda do laboratório. Com uma máquina de microfluídica, cada célula foi individualizada, seu RNA convertido em DNA e etiquetado para identificação.
Esse processo, chamado sequenciamento de RNA de célula única, permitiu visualizar a expressão gênica em milhares de células simultaneamente. O professor David Jackson explicou:
“A grande vantagem é que agora existe um atlas de expressão gênica. Quando publicarmos esse material, toda a comunidade poderá utilizá-lo. Outros pesquisadores interessados em células-tronco de milho ou Arabidopsis não precisarão repetir o experimento. Eles poderão usar nossos dados.”
(“The great thing is that you have this atlas of gene expression. When we publish that, the whole community can use it. Other people interested in maize or Arabidopsis stem cells don’t have to repeat the experiment. They will be able to use our data.”)— David Jackson, Professor, Cold Spring Harbor Laboratory
O sequenciamento possibilitou a recuperação de cerca de 5.000 células expressando CLAVATA3 e 1.000 com WUSCHEL. Foram identificados ainda centenas de genes expressos preferencialmente em células-tronco das duas espécies, indicando importante papel evolutivo. O estudo também associou reguladores específicos de células-tronco à produtividade no milho, perspectiva relevante para seleção agrícola.
Implicações e Próximos Passos
A identificação de reguladores-chave dessas células cria as bases para inovação em biotecnologia agrícola, regeneração de plantas e aumento de produtividade. “Idealmente, gostaríamos de saber como fabricar uma célula-tronco. Isso nos permitiria regenerar plantas de maneira mais eficiente e entender a diversidade vegetal”, afirmou Jackson:
“Ainda não temos uma lista completa dos reguladores — os genes necessários para isso.”
(“We don’t yet have a full list of regulators — the genes we need to do that.”)— David Jackson, Professor, Cold Spring Harbor Laboratory
Com esse novo atlas, cientistas do mundo inteiro poderão explorar dados sem a necessidade de replicar experimentos complexos, acelerando avanços na engenharia genética de culturas. Além de orientar estudos básicos sobre desenvolvimento vegetal, a descoberta deve impactar diretamente áreas como fisiologia agrícola e programas de melhoramento, contribuindo para novas estratégias de produção de alimentos e energia renovável.
A expectativa é que, nos próximos anos, esses dados sirvam de referência para investigações sobre diversidade e adaptação de plantas, aproximando a ciência de soluções eficazes ao desafio alimentar global.
Fonte: (ScienceDaily – Ciência)





